Estado da Arte
Inibidores de Proteases Oriundas de Plantas: Uma Abordagem Útil para o Desenvolvimento de Novos Fármacos
Proteases Inhibitors Originated from Plants: Useful Approach for Development of New Drug
Resumo
As proteases e seus inibidores estão amplamente distribuídos em todos os organismos vivos. São enzimas que desempenham funções crucias para a manutenção da vida e sua atividade é mantida sob estrito controle por mecanismos que incluem os inibidores e, se esta atividade não for controlada, pode acarretar doenças e até mesmo a morte do organismo. Os inibidores de ocorrência natural são peptídeos de tamanho variável e são classificados de acordo com o tipo de proteases que inibem. Os inibidores de serino-proteases são os mais importantes da natureza e também os mais expressos em plantas, e será o foco desta revisão. São classificados como inibidores canônicos, não-canônicos e Serpinas, de acordo com a estrutura e mecanismo de ação. As plantas expressam estes inibidores constitutivamente ou são induzidos por algum agente externo. Estes inibidores aumentam a resistência da planta a parasitos, insetos, larvas, microorganismos, pragas e patógenos em geral. São expressos geralmente nas sementes e grãos e levam a morte ou repelem o organismo invasor. Diversas pesquisas estão sendo conduzidas para purificar, caracterizar e avaliar o efeito de inibidores de serino-proteases em plantas no tratamento de diversas patologias humanas, inclusive alguns inibidores, como no caso dos inibidores de BBI de soja, já estão em estudos clínicos de fase II.
- Unitermos:
- Inibidores de Proteases de Plantas; Peptídeos.
- Serino-Proteases.
Abstract
Proteases and their inhibitors are found in all living organisms. These enzymes have crucial functions and their activities are regulated by many mechanisms, including inhibitors, but if their activities are not under control they can develop diseases and ultimately death. Natural inhibitors are variable sized peptides and are classified by the inhibited protease type. Serine protease inhibitors are among the major and most important inhibitors expressed in plants, and will be the focus of this revision. They are classified as: canonical inhibitors, non-canonical and Serpins, according to their structure and mechanism of action. Plants can express inhibitors constitutively or are induced by external stimuli and they confer resistance against parasites, insects, larval, microorganisms, plagues and pathogens. They are mainly expressed in seeds and grains, killing or repelling invader organisms. Many investigations are been conducted in order to purify, characterize the plant serine protease inhibitors and to evaluate their effects on the treatment of a large number of human pathologies. In addition, some inhibitors have already been tested in phase II clinical studies, such as soybean BBI.
- Key Words:
- Protease Inhibitors; Peptides.
- Serine Protease.
Introdução
As proteases, melhor denominadas de peptidases, catalisam reações de hidrólise de diferentes ligações peptídicas em proteínas e em peptídeos de todos os organismos vivos. Tais reações estão envolvidas em processos fisiológicos de extrema importância para a sobrevivência destes organismos, tais funções incluem: digestão e assimilação das moléculas alimentares, degradação de protéinas nos lisosomas e nos proteasomas, cascatas de coagulação, complemento e as de sinalização intracelular, fibrinólise, degradação de moléculas da matriz extracelular, dentre outros processos (NEURATH, 1989). Além da sua relevância fisiológica, as proteases podem ser potencialmente nocivas para o ambiente protéico de uma célula ou de um organismo, logo suas atividades requerem um controle preciso, visto que a hidrólise de uma ligação peptídica é um evento irreversível. Quando a atividade das proteases torna-se descontrolada ou esta controlada de maneira inapropriada, processos patológicos podem se instalar num organismo e até mesmo levá-lo à morte. Enfermidades como enfisema pulmonar, epilepsia, síndrome de Netherton, doenças hepáticas, cânceres, doenças reumáticas, doenças degenerativas, doenças auto-imunes, dentre outras estão relacionadas a um descontrole da atividade das enzimas proteolíticas (BERG, 2008; HEAL et al., 2008; KUESTER et al., 2008). O nível básico de controle da atividade de uma protease é a regulação da sua expressão gênica, da sua secreção, da sua síntese sob forma de zimogênio - que é um precursor inativo com maior massa molecular do que a protease ativa - ou ainda, o direcionamento da enzima para sua degradação. Já o segundo nível de regulação, e o mais importante, é a inibição da atividade através da interação direta da protease com inibidores específicos (KROWARSCH et al., 2003). Estes inibidores, sintéticos ou naturais, têm sido utilizados, por administração oral, com grande êxito no tratamento de algumas enfermidades. A excessiva proteólise observada em um número de condições, como as citadas anteriormente, pode ser tratada por terapia gênica, introduzindo-se a maquinaria gênica para a síntese do inibidor da referida atividade proteolítica. No campo da agricultura, algumas plantas geneticamente modificadas, expressando inibidores de enzimas proteolíticas digestivas de insetos, pragas e pestes, têm sido introduzidas em culturas; e os resultados obtidos apontam para um aumento considerável na resistência destas plantas aos agentes agressores em questão (RAWLINGS et al., 2004).
Inibidores de Proteases
Os inibidores de proteases podem ser genericamente classificados de acordo com sua estrutura em dois grandes grupos: inibidores de baixo peso molecular e inibidores peptídicos com uma ou mais cadeias polipeptídicas. Quase todos os inibidores de proteases de ocorrência natural são polipeptídeos; apenas alguns microorganismos secretam pequenos compostos não-peptídicos que bloqueiam a atividade das proteases de seus hospedeiros (POWERS ET AL., 2002). Estes inibidores ainda podem ser classificados de acordo com o tipo de protease que inibem: inibidores de serino, cisteíno, aspártico, treonino e metaloproteases. É bem verdade que alguns inibidores interferem com mais de um tipo catalíco de protease com diferentes afinidades de ligação, ou seja, possuem distintos valores de constantes de inibição (Ki) para os diferentes tipos de proteases (FEAR et al., 2007). Um exemplo deste comportamento é observado para o inibidor do tipo Kunitz encontrado em anêmonas do mar da espécie Stichodactyla helianthus, que é um potentíssimo inibidor da atividade de serino-proteases, com valores de Ki entre 10-9 a 10-7M, embora iniba outros tipos de peptidases com menor afinidade de ligação, ou seja, apresentam maiores valores de Ki do que aqueles observados para as serino-proteases (DELFÍN et al., 1996). Este inibidor demonstrou expressivo efeito inibitório na atividade das serino-proteases de Leishmania amazonensis, e em promastigotas de cultura induziu importantes alterações morfológicas e a formação de vacúolos autofágicos, o que levou o parasita à morte, fazendo deste peptídeo uma molécula com grande potencial na terapêutica anti-Leishmania e atribuindo às suas serino-proteases importantes alvos terapêuticos (SILVA-LOPEZ et al., 2007).
As maneiras pelas quais os inibidores interagem com suas enzimas-alvo variam enormemente, mas existem dois mecanismos gerais e distintos de inibição enzimática representados pela inibição irreversível e pela inibição reversível de forte ligação.
A inibição irreversível é específica para as endopeptidases, pois é dependente da clivagem enzimática de uma ligação peptídica no interior da cadeia do inibidor. Este então se liga covalentemente à enzima, disparando uma importante alteração na sua estrutura tridimensional, que leva ao desalinhamento dos aminoácidos catalíticos do sítio ativo da peptidase levando assim a sua desnaturação (LASKOWSKI; KATO, 1980). Este tipo de cinética, ou seja, a clivagem de uma ligação peptídica do inibidor, é exclusiva para os inibidores peptídicos, que é o grupo mais importante, mas existem também aqueles inibidores de baixo peso molecular que não sofrem hidrólise e se ligam a diferentes sítios da enzima levando a desnaturação da mesma. As três famílias de inibidores que exibem este tipo de cinética de inibição são I4, I39 e I50 (Tabela 1). A maioria dos inibidores de ocorrência natural utiliza o mecanismo de inibição reversível de forte ligação, pois interagem com grande afinidade com o sítio ativo da protease de um modo semelhante ao da ligação enzima-substrato. Este inibidor pode ser clivado ou não durante o processo de inibição, e ser liberado do sítio ativo da enzima não modificado ou íntegro. Este mecanismo padrão de inibição tem sido demonstrado conclusivamente apenas para as serino-proteases. Foram reconhecidas 19 famílias de inibidores de acordo com este padrão de inibição (LASKOWSKI; QASIM, 2000). A Figura 1 esquematiza a interação dos aminoácidos catalíticos (S) do sítio ativo da enzima com os aminoácidos que sofrerão hidrólise no peptídeo (P). É importante ressaltar que a clivagem da ligação peptídica ocorre entre os aminoácidos P1 e P1´ (SCHECHTER; BERGER, 1967).
| Família | Nome | Inibidor representante típico | Família de peptidase inibida* |
| I1 | Kazal | Unidade 3 ovomucóide (Meleagris gallopavo) | S1 |
| I2 | Kunitz (animal) | Aprotinina (Bos taurus) | S1 |
| I3A§ | Kunitz (vegetal) | Inibidor triptico da soja (Glycine max) | S1, C1 e A1 |
| I3B | Inibidor de protease B (Sagitaria sagittilolia) | S1 | |
| I4 | Serpina | α 1-antitripsina (Homo sapiens) | S1, S8, C1 e C14 |
| I5 | Ascidiana | Inibidor tríptico de ascidia (Halocynthia roretzi) | S1, |
| I6 | Cereal | Inibidor de α amilase (Eleusine coracana) | S1 |
| I7 | Squash | MCTI-1 inibidor tríptico (Momordica charantia) | S1 |
| I8 | Ascaris | Inibidor anticoagulante nematoda (Ascaris suum) | S1 e M4 |
| I9 | YIB | Inibidor de protease B (Saccharomyces cerevisiae) | S8 |
| I10 | Marinostatina | Marinostatina (Alteromonas sp) | S1 |
| I11 | Ecotina | Ecotina (Escherichia coli) | S1 |
| I12 | Bowman-Birk | Inibidor tríptico de planta (Glycine max) | S1 e C1 |
| I13 | Pot 1 | Eglina C (Hirudo medicinalis) | S1 e S8 |
| I14 | Hirudina | Hirudina (Hirudo medicinalis) | S1 |
| I15 | Antistatina | Antistatina unidade 1 (Haementeria officinalis) | S1 |
| I16 | SSI | Inibidor subtilisina (Streptomyces albogriseolus) | S1 |
| I17 | Elafina | Inibidor de proteinase do muco (Homo sapiens) | S1 |
| I18 | Mostarda | Inibidor tríptico da mustarda (Sinapis alba) | S1 |
| I19 | Pacifascina | Inibidor de proteinase LCMI (Locusta migratoria) | S1 |
| I20 | Pot2 | Inibidor de proteinase II (Solanum tuberosum) | S1 |
| I21 | 7B2 | Secretogranina V (Homo sapiens) | S8 |
| I24 | pinA | Inibidor de endopeptidase La (bacteriofago T4) | S16 |
| I25A | Cistatina 1 | Cistanina A (Homo sapiens) | C1 |
| I25B | Cistaina 2 | Ovocistatina (Gallus gallus) | C1 e C13 |
| I25C | Cistatina 3 | Inibidor de metaloproteases (Bothropus jararaca) | M12 e S8 |
| I27 | Calpastatina | Calpastatina 1 (Homo sapiens) | C2 |
| I29 | Antígeno α2 T citotóxico (Mus musculus) | ||
| I31 | Tiropina | Equistatina (Actinia equina) | C1 |
| I32 | IAP | Proteína BIRC (Homo sapiens) | C14 |
| I33 | Ascaris PI3 | Inibidor PI3 de pepsina (Ascaris suum) | A1 |
| I34 | IA3 | Inibidor sacaropepsina (Saccharomyces cerevisiae) | A1 |
| I35 | Timp | Timp-1 (Homo sapiens) | M10 e M12 |
| I36 | SMI | Inibidor de metaloprotease (Streptomyces nigrescens) | M4 |
| I37 | PCI | Inibidor de carboxipeptidase do tomate (Solanum tuberosum) | M14 |
| I38 | Aprina | Inibidor de metaloprotease (Erwinia chrysanthemi) | M10 |
| I39 | α2M | α2-macroglobulina (Homo sapiens) | Várias S, C, M e A |
| I40 | Bombyx | Inibidor de subtilisina bombyx (bombyx mori) | S8 |
| I42 | Chagasina | Chagasina (Trypanosoma cruzi) | C1 |
| I43 | Oprina | Oprina (Didelphis marsupialis) | M12 |
| I44 | ICA | Inibidor de carboxipeptidaseA (Ascaris suum) | M14 |
| I46 | LCI | Inibidor de carboxipeptidase (Hirudo medicinalis) | M14 |
| I47 | Latexina | Latexina (Homo sapiens) | M14 |
| I48 | Clitocipina | Clitocipina (Lepista nebularis) | C1 |
| I49 | proSAAS | proSAAS (Homo sapiens) | S8 |
| I50 | p35 | Inibidor de caspase p53 (Spodoptera litura) | C14 |
| I51 | IC | Inibidor de carboxipeptidase Y (Saccharomyces cerevisiae) | S10 |
| I52 | TAP | Peptídeo anticoagulante (Ornithodonus moubata) | S1 |
| I53 | Madanina | Madanina 1 (Haemaphysalis longicornis) | S11 |
| I57 | Estafostatina B (Staphylococcus aureus) | C1 | |
| I58 | Estafostatina A (Staphylococcus aureus) | C1 | |
| I59 | Triabina | Triabina (Triatoma pallidipennis) | S1 |
| I63 | Proteína básica de eosinófilo (Homo sapiens) | S3 | |
| I64 | Trombostasina | Trombostasina (Haematobia irritans) | S1 |
| I67 | Bromeina | Bromeína (Ananas comosus) | C1 |
| I68 | Inibidor de Carboxypeptidase (Rhipicephalus bursa) | M10 | |
| I71 | Falstatina | Falstatina (Plasmodium falciparum) | C13 |
| I72 | Chimadanina | Chimadanina (Haemaphysalis longicornis) | S1 |
| I73 | Inibidor tríptico de Veronica (Veronica hederifolia) | S1 | |
| I74 | Variegina (Amblyomma variegatum) | S8 | |
| I75 | Proteína CIII (“bacteriophage lambda”) | A1 | |
| I76 | Inibidor de Trombin (Glossina morsitans) | S1 | |
| I77 | Anofelina | Anofelina (Anopheles albimanus) | S1 |
| I78 | Inibitor de elastase (Aspergillus fumigatus) | S1 | |

A importância dos inibidores de protease
As pesquisas na área de inibidores de proteases têm gerado um grande e rápido fluxo de informações, mas o grande problema de lidar com tais informações reside na enorme dificuldade de nomenclatura deste grupo tão diversificado de substâncias. Tais inibidores são geralmente descobertos pela sua atividade inibitória contra enzimas que estão disponíveis no mercado. Entre elas, as mais comuns são tripsina, quimiotripsina ou subtilisina, que são serino-proteases; e com isso os inibidores passam a denominar-se de acordo com o organismo ou o tecido de origem, como por exemplo: inibidor de subtilina de Streptomyces ou inibidor de tripsina pancreático. Tais denominações não oferecem informações relevantes sobre a relação entre os inibidores e ainda torna difícil o esclarecimento sobre os aspectos do seu mecanismo de ação. Portanto, as informações sobre a estrutura destes inibidores é essencial para entender melhor sua atividade, bem como para sua classificação.
Através de um estudo minucioso e inovador, com o objetivo de melhor organizar os inibidores de proteases e propor uma nomenclatura mais adequada, Rawlings e colaboradores utilizaram bancos de dados de estruturas primárias - seqüências de aminoácidos e a localização das pontes de enxofre de proteínas e peptídeos - apoiados em algoritmos computacionais. Por intermédio deste estudo, os inibidores foram agrupados em 48 famílias, de acordo com a similaridade de sequência, estrutura tridimensional, bem como do mecanismo específico de inibição. Atualmente, esta lista foi aumentada para 68 famílias segundo o banco de dados MEROPS (versão 7.6), contém informações de proteases, suas estruturas, características cinéticas e bioquímicas; assim como seus inibidores (RAWLINGS; MORTON, 2008). A Tabela 1 é uma compilação de todas as famílias de inibidores de proteases com seqüências peptídicas depositadas em bancos de dados, como SWISSPROT, TrEMBL e PDB.
Uma família é um conjunto de inibidores com sequências homólogas e esta é dada por uma significante similaridade da estrutura primária com um exemplar de uma família. Uma família pode conter um ou vários inibidores, e um simples produto gênico pode possuir diversos domínios inibitórios. Cada família é denominada pela letra “I” e pode ainda ser dividida em subfamílias quando existir ancestral divergente na família. O representante mais característico de cada família é o que dá o nome a ela; este inibidor é via de regra o melhor caracterizado bioquímica, cinética e estruturalmente, e geralmente é o primeiro membro descoberto (RAWLINGS; MORTON, 2008). Estas famílias são ainda agrupadas em grandes clãs. O clã designa uma linha evolucionária de inibidores que é definida pelo tipo de enovelamento. Um clã contém uma ou mais famílias completas, desde que todos os membros destas famílias assumam o mesmo tipo de estrutura tridimensional (RAWLINGS; BARRET, 1993). A maior família de inibidores são as Serpinas (I4) com mais de 500 inibidores com sequências determinadas, resolvidas e depositadas em banco de dados, Por outro lado existem famílias como I5, I24, I34, I36, I40, I44 e I46, que são representadas por apenas um único inibidor (RAWLINGS et al., 2004).
Inibidores de proteases de plantas
Os inibidores de proteases de plantas são polipeptídeos de baixo peso molecular, com a capacidade de inibir uma grande variedade de enzimas proteolíticas, incluindo proteases digestivas de mamíferos, insetos, bactérias e fungos, e estão também envolvidos na regulação do metabolismo de proteínas endógenas das plantas (CLEMENTE; DOMONEY, 2006). Os inibidores de proteases exibem importantes funções na regulação da proteólise em diversos organismos e em plantas de modo especial, constituindo importantes estratégias de defesa contra predadores e patógenos, visto que as plantas não possuem um sistema imune (ZHANG et al., 2008). Tais inibidores podem ser sintetizados constitutivamente ou a síntese ser induzida em resposta ao ataque de agentes externos. Estes inibidores peptídicos de proteases são encontrados em plantas de vários grupos sistemáticos, com particular ênfase nas espécies da família Fabaceae (uma das maiores famílias botânicas e mais conhecidas como Leguminosae). A maioria dos inibidores é estável à variação de temperatura e de pH; e além de sua função biológica natural, podem ser utilizados no tratamento de determinadas patologias (YOSHIZAKI et al., 2007).
Inibidores de serino-proteases
É importante chamar atenção sobre o fato das serino-proteases serem as mais abundantes e as mais bem estudadas enzimas proteolíticas da natureza; conseqüentemente seus inibidores também o são. Como se pode observar na Tabela 1, aproximadamente 57 % das famílias de inibidores atuam positivamente sobre as serinoproteases. Tais inibidores peptídicos apresentam estruturas distintas e podem inibir suas enzimas alvo tanto por inibição reversível, que é o mecanismo mais comum observado; quanto por inibição irreversível, e são ainda classificados como inibidores canônicos, não-canônicos e Serpinas (KROWARSCH et al., 2003). Os inibidores de plantas de outras classes de proteases não são menos importantes, mas aparentemente são menos numerosos e não estão tão bem caracterizados quanto os inibidores de serino-proteases. De um modo geral, as plantas expressam de maneira significativa quantidades muito maiores de inibidores de serino-proteases do que os outros inibidores, e por este motivo os inibidores de outras classes de proteases não serão discutidos nesta revisão.
Os inibidores canônicos representam o maior grupo de inibidores de serino-proteases e estão distribuídos essencialmente em todos os grupos de organismos, compreendendo polipeptídeos de 14 a 200 aminoácidos. Não formam um simples grupo de moléculas, mas são dividos em várias famílias, ou seja, várias famílias constituem inibidores do tipo canônico. Em plantas, eles se acumulam em grandes quantidades nas sementes. Estes inibidores apresentam como característica cinética marcante uma ligação forte, mas não covalente com o sítio ativo da protease, não induzindo alteração na estrutura tridimensional da enzima. Esta ligação se assemelha ao complexo de Michaelis na interação enzima-substrato (OTLEWSKI, et al, 2005). O sítio reativo do inibidor tem a ligação peptídica P1-P1’ localizada numa curvatura externa (loop) do peptídeo, denominado de RCL (Reactive Center Loop), e a reação de inibição se processa em pH alcalino, que é o pH ótimo das serino-proteases. Embora a estrutura tridimensional dos inibidores como um todo seja diferente, a região de ligação com a enzima (ou seja, a região de inibição) é quase idêntica e, portanto, o mecanismo de ação é muitíssimo parecido. As famílias de inibidores de serino-proteases canônicos de plantas incluem: Kazal, Tomate 1 e 2, Cereal, Inibidor de tripsina da soja, Squash, Bowman-Birk, Batata 1 e 2, Semente de Colza (KROWARSCH et al., 2003). Existem inibidores de plantas que por conta da sua estrutura primária e tridimensional podem estar alocados em famílias juntamente com inibidores de outros organismos.
Inibidores da família Bowman-Birk (BBI) podem ser encontrados em muitas famílias botânicas, mas são particularmente abundantes nas sementes de leguminosas, quando comparados com os outros orgãos do legume e também com outras famílias de plantas (CLEMENTE; DOMONEY, 2006). São também abundantes na família Poaceae (gramíneas como trigo, aveia, cevada, arroz, milho e canade-açúcar) (QI et al., 2005). Seus representantes mais importantes e mais bem estudados são encontrados nas sementes de soja (Glycine max) e de ervilha (Pisum sativum) e inibem fortemente a atividade de diversas tripsinas e quimiotripsinas (LOSSO, 2008). A síntese destes é induzida por infecções ou por lesões, sugerindo um amplo papel na defesa e na resistência nas plantas contra pestes e patógenos. Estes inibidores são estáveis à temperatura de cozimento e também aos valores de pH do sistema digestivo humano e de outros animais. Isto se deve à presença das sete pontes de enxofre na sua estrutura primária (Figura 2) e, por conta desta grande estabilidade, os BBI demonstram possuir grande potencial de aplicação clínica (QI et al., 2005). São utilizados em plantas transgênicas para conferir defesa contra insetos e para o tratamento de enfermidades humanas. As larvas de Diatraea saccharalis alimentam-se das folhas de cana-de-açúcar e crescem sobre ela. Experimentalmente, quando se introduzem os genes do BBI e Kunitz da soja nesta gramínea, o crescimento da larva é drasticamente reduzido, e a produção de cana bastante aumentada (FALCO; SILVA-FILHO, 2003).
Os BBI de leguminosas possuem uma importante atividade anticarcinogênica e radioprotetora. Adicionalmente, tem sido observado que as populações com dietas ricas em leguminosas e em gramíneas apresentam baixa incidência de cânceres de cólon, próstata, orofarígeo, mama e pele (KENNEDY, 1998). Este efeito possivelmente é resultante da inibição da proteólise provocada pelas células transformadas já que necessitam das proteases para os eventos de metástase, angiogênese, crescimento tumoral e proliferação (DELL’AICA, 2007). Estudos in vitro têm demonstrado que os BBI, em concentrações nanomolares, são capazes de suprimir a transformação de células malignas induzidas por raios-X, de inibir a produção do radical livre ânion superóxido por células humanas leucêmicas de linhagem de pro-mielócito, de potencializar a morte induzida por radiação e cisplatina em vários cânceres humanos como de mama, cérvix, cabeça, pescoço, pulmão, de suprimir o crescimento de tumores enxertados em camundongos, de bloquear a tumorigênese induzida por estrogênios em células de mama, dentre outros efeitos em outras linhagens tumorias e em tumores sólidos (CLEMENTE; DOMONEY, 2006). Estudos in vivo mostraram que os BBI previnem e suprimem o desenvolvimento de cânceres como adenocarcinoma anal e de cólon, esôfago, pulmão, linfosarcoma e carcinoma oral, em diferentes modelos animais induzidos por distintos agentes carcinogênicos (LOSSO, 2008). É importante ressaltar que muitos BBI naturais ou modificados como seqüências ativas do BBI, resultantes da hidrólise da sua estrutura primária, como os loops que se ligam diretamente à enzima, já se encontram em estudos clínicos de Fase I e II para o tratamento de vários tipos de neoplasias (CLEMENTE; DOMONEY, 2006; LOSSO, 2008). Os BBIs também demonstraram grande potencial de aplicação em dengue, pois foi reportado um importante efeito inibitório na protease viral NS3, essencial na replicação do vírus do Dengue (SAMPATH; PADMANABLAN, 2008). Além disso, os BBIs e seus respectivos esqueletos canônicos (porção estrutural do inibidor responsável pela inibição; ver Figura 2) são inibidores potentíssimos da atividade das serino-proteases humanas Triptase, Matriptase e Elastase leucocitária - que são enzimas envolvidas em várias desordens inflamatórias, alérgicas e degenerativas como asma, esclerose múltipla, artrite reumatóide, enfisema pulmonar, fibrose cística, bronquite e síndromes respiratórias (QI et al., 2005).

Outra importante família de inibidores canônicos de plantas, isolados inicialmente em espécies da família Cucurbitaceae e com promissor potencial terapêutico é a família de inibidores Squash. São polipeptídeos pequenos, com 27 a 34 aminoácidos e seis resíduos de cisteínas envolvidas em três pontes de enxofre, formando três alças peptídicas de tamanhos variados, resultando assim numa estrutura tridimensional com aspecto de laços (Figura 3). É importante destacar que existem pequenas proteínas denominadas knotinas ou ciclotinas que compartilham semelhanças estruturais com os inibidores squash devido à topologia das suas pontes de enxofre e sua estrutura tridimensional em laços, e é bem provável que em plantas, estes inibidores sejam provenientes do metabolismo destas knotinas (CHICHE et al., 2004). Os inibidores Squash apresentam constantes de associação com várias serino-proteases na ordem de 10-12 M e, portanto, são considerados os mais potentes inibidores de proteases da natureza. Na planta, estes inibidores participam de mecanismos envolvidos na defesa conferindo resistência a pragas e pestes (OTLEWSKI; KROWARSCH, 1996). A estrutura “macrociclitizada”, por conta das pontes de enxofre, confere a estes inibidores uma grande resistência à ação das exopeptidases e também a altas temperaturas, possibilitando assim, sua administração oral e a permanência de suas atividades inibidoras de proteases (GÖRANSSON et al., 2004). São potentes inibidores de serinoproteases de interesse médico como a Elastase de neutrófilo envolvida na patogênese de diversas enfermidades como enfisema, fibrose cística e artrite reumatóide, a Trombina também denominada de Fator II da cascata de coagulação, e a enzima Matriptase, envolvida na destruição tecidual observada em vários tipos de neoplasias (CRAIK et al., 2004). Os representantes mais estudados desta família são MCoTI-I e II, ambos isolados das sementes de Monocardia cochinchinensis, uma Cucurbitaceae comum do Vietnã. Diversos estudos estão sendo conduzidos no sentido de modificar a estrutura primária destes inibidores por mutagênese induzida e, por modelagem molecular induzir a obtenção de inibidores mais potentes e mais específicos, reforçando assim a idéia de que estes peptídeos são moléculas promissoras no desenvolvimento racional de fármacos para o tratamento de inúmeras patologias humanas (CHICHE ET AL., 2004).

Os inibidores não-canônicos exibem uma estrita especificidade para uma determinada protease, pois, além da ligação do inibidor com a enzima assemelhar-se ao complexo enzima-substrato (como observado para os inibidores canônicos), o inibidor também se liga a um sítio na superfície da protease, aumentado muitíssimo a seletividade da inibição (FARADY et al., 2008). Este tipo de inibidor está envolvido na regulação de mecanismos como apoptose, sinalização intracelular, embriogênese, angiogênese, neurogênese e em eventos relacionados à resposta imune (WOODS et al., 2008). Até o momento, ainda não foi reportado nenhum tipo de inibidores não-canônicos em plantas; contudo as investigações sobre estes peptídeos são bastante recentes. É importante destacar que os inibidores não-canônicos apresentam um enorme potencial de aplicabilidade no tratamento de determinadas patologias devido à altíssima especificidade que demonstram.
As Serpinas compreendem uma superfamília de proteínas com estruturas muito semelhantes, e são inibidoras de serino-proteases, de onde seu nome deriva (serine protease inhibitors). Os primeiros representantes estudados foram a antitrombina e a antitripsina, que são proteínas plasmáticas essenciais na regulação das cascatas de coagulação e de inflamação e suas deficiências resultam em tromboses e em enfisema pulmonar, respectivamente (RAWLINGS et al., 2004). Enquanto que a grande maioria das Serpinas controla cascatas proteolíticas, outras não inibem a atividade enzimática, mas exercem uma diversidade de funções. São armazenadoras de proteínas, como a ovalbumina; proteínas de transporte de hôrmonios, como as globulinas de ligação da tiroxina e do cortisol; ou são supressoras de genes tumorais com a maspina (ROBERTS; HEJGAARD, 2008). São proteínas de médio tamanho (cerca de 400 aminoácidos), cuja estrutura terciária consiste de três β-hélices (A, B e C), nove α-hélices e um loop central reativo que é exposto na superfície do inibidor (RCL), com cerca de 25 resíduos entre as A e C (Figura 4). Ao reagir irreversivelmente com o sítio ativo da enzima através do RCL, a enzima cliva o inibidor e esta clivagem induz uma profunda alteração conformacional tanto do inibidor quanto da enzima. De um modo geral, as Serpinas são inibidoras irreversíveis das serino-proteases da família quimiotripsina, mas outras enzimas são também inibidas como tripsina, trombina, elastase, fatores da coagulação, e também algumas cisteíno-proteases (OTLEWSKI et al., 2005).
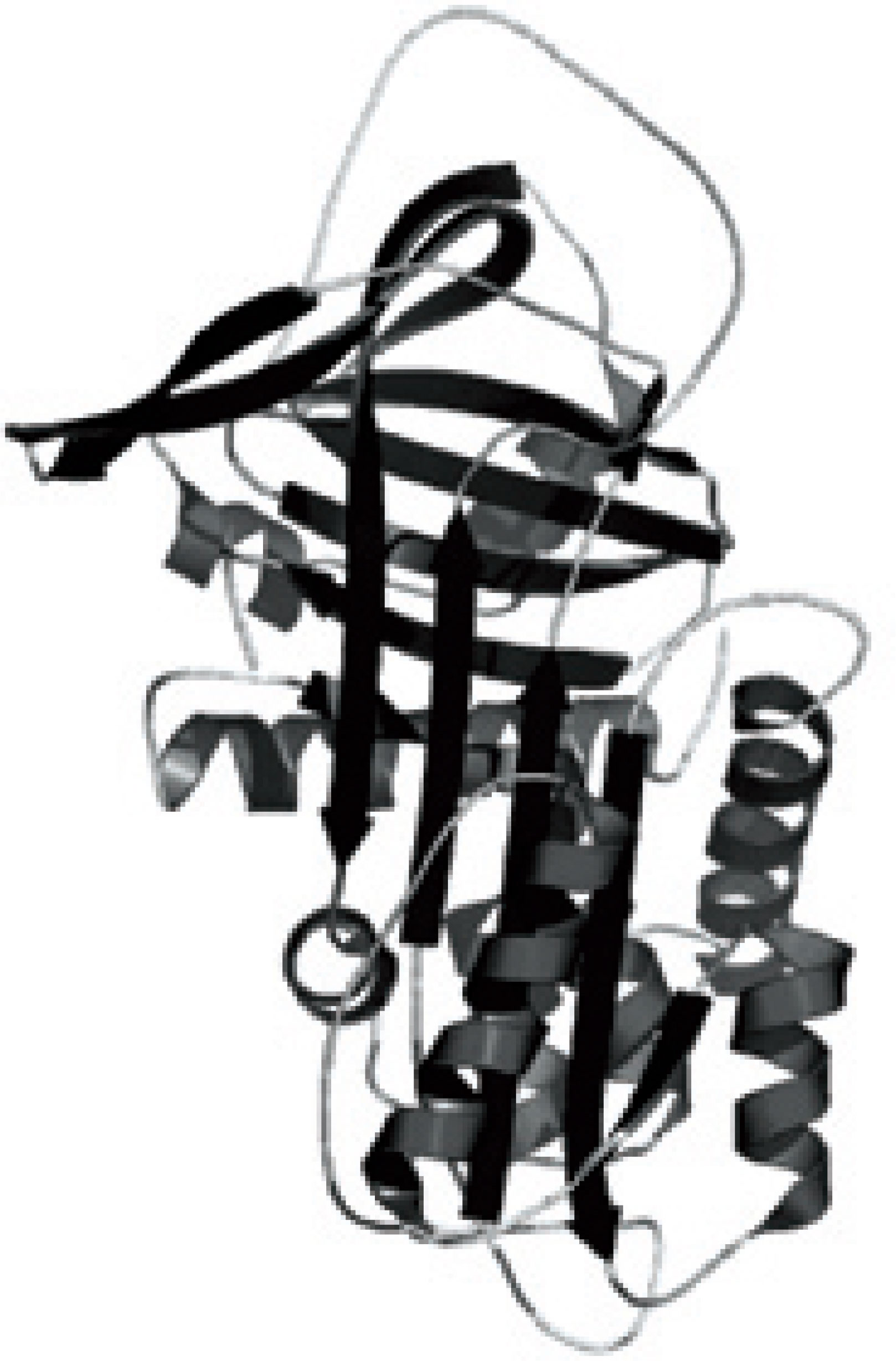
As Serpinas estão distribuídas em animais, plantas, bactérias, archaea, e em certos vírus, e constitui a maior família de inibidores de proteases. A primeira Serpina estudada em plantas, foi uma proteína de 43 kDa, denominada de proteína Z, obtida de grãos de cevada. Diversas Serpinas foram purificadas de outras gramíneas como aveia, arroz, trigo, milho, cana e bambu. Embora sejam expressas principalmente em gramíneas, são também encontradas na soja, algodão, tomate, maça dentre outros (ROBERTS; HEJGAARD, 2008). Como são particularmente abundantes nas sementes, aparentemente desempenham um importante papel na defesa do estoque protéico contra as enzimas digestivas de insetos e fungos. Genes que codificam Serpinas têm sido introduzidos em culturas de alfafa, batata, algodão e tabaco, aumentando significativamente a resistência destes ao ataque de insetos e predadores (CHRISTELLER; LAING, 2005). Estes inibidores garantem a integridade de peptídeos sinalizadores envolvidos na apoptose, que é a morte celular não seguida de autólise que ocorre de forma ordenada e demanda energia para a sua execução. Está relacionada ao equilíbrio interno dos organismos e com sua regulação fisiológica. O termo é derivado do grego, que se referia à queda das folhas das árvores no outono - um exemplo de morte programada fisiológica (WILLIAMS; DICKMAN, 2008). É importante destacar que a apoptose participa da diferenciação dos órgãos da planta, desde a senescência à resposta ao estresse como hipóxia, temperaturas extremas e oxidação (ROBERTS; HEJGAARD, 2008). As Serpinas de origem humana e de outros animais têm sido utilizadas com grande sucesso na terapêutica de muitas patologias. Plantas transgênicas que carregam genes que codificam Serpinas são amplamente utilizadas na agricultura, mas apenas recentemente os estudos de Serpinas de plantas apontam para uma aplicabilidade clínica. Um exemplo disto é a expressão de um Serpina de um arroz trangênico que se mostrou bastante efetiva na redução e no controle da pressão arterial (YANG et al., 2006).
Considerações finais
As proteases desempenham funções vitais nas plantas e seus inibidores também participam destes processos regulando suas atividades. Além disso, estes inibidores protegem as plantas de modo bastante eficiente do ataque de agentes externos, como parasitos, insetos, larvas e microorganismos, inibindo preferencialmente suas enzimas digestivas. Devido à alta especificidade, estabilidade aos extremos de pH, temperatura e alta hidrossolubilidade, estes inibidores são facilmente administrados oralmente. A observação de que são eficientes contra microorganismos e parasitos, apontam estes inibidores de proteases de plantas com um enorme potencial para o desenvolvimento de novos fármacos, sugerindo uma maior eficiência e efeitos adversos menos agressivos para o hospedeiro. No entanto, as pesquisas sobre os inibidores de proteases provenientes de plantas são relativamente recentes, e é necessário que as investigações nesta área sejam intensificadas para a melhor compreensão destas estruturas. O entendimento do funcionamento destas moléculas com certeza tem muito a contribuir com o desenvolvimento racional de medicamento.
Referências
BERG, T. Small-molecule inhibitors of protein-protein interactions. Current Opinion in Drug Discovery Development, v.11 p.666-74, 2008.
CHICHE, L.; HEITZ, A.; GELLY, J.C.; GRACY, J.; CHAU, P.T.T.; HA, P.T.; HERNANDEZ, J.F.; LENGUYEN, D. Squash inhibitors: From structural motifs to macrocyclic knottins. Current Protein Peptide Scence, v.5, p.341-349, 2004.
CHRISTELLER, J.T.; LAING, W. Plant serine proteinase inhibitors. Protein Peptide Letter, v.12, p.439-447, 2005.
CHRISTMANN, A.; WALTER, K.; WENTZEL, A.; KRÄTZNER, R.; KOLMAR, H. The cystine knot of a squash-type protease inhibitor as a structural scaffold for Escherichia coli cell surface display of conformationally constrained peptides. Protein Engineering, v.12, p.797-806, 1999.
CLEMENTE, A.; DOMONEY, C. Biological significance polymorphism in legume protease inhibitor the Bowman-Birk family. Current Protein Peptide Scence, v.7, p.201-216, 2006.
CRAIK, D.J.; DALY, N.L.; MULVENNA J.; PLAN, M.R.; TRABI, M. Discovery, structure and biological activities of the cyclotides. Current Protein Peptide Scence, v.5, p.297-315, 2004.
DELFÍN, J.; MARTÍNEZ, I.;ANTUCH, W.; MOREIRA, V.; GONZALÉZ, Y.; RODRÍGUEZ, R.; MÁRQUEZ, M.; SAROYÁN, A.; LARIONOVA, N.; DÍAZ, J.; PADRÓN, G.; CHÁVEZ, M. Purification, characterization and immobilization of proteinase inhibitors from Stichodactyla helianthus. Toxicon, v.34, p.1367-76, 1996.
DELL’AICA, I.; CANIATO, R.; BIGGIN, S.; GARBISA, S. Matrix proteases, green tea, and St. John’s wort: Biomedical research catches up with folk medicine. Clinical Chimical Acta, v.381, p.69-77, 2007.
FALCO, M.C.; SILVA-FILHO, M.C. Expression of soybean proteinase inhibitors in transgenic sugarcane plants: Effects on natural defense against Diatraea saccharalis. Plant Physiology Biochemistry, v.41, p.761-766, 2003.
FARADY, C.J.; EGEA, P.F.; SCHNEIDER, E.L.; DARRAGH, M.R.; CRAIK, C.S. Structure of an Fab-protease complex reveals a highly specific noncanonical mechanism of inhibition. Journal of Molecular Biology, v.380, p.351-360, 2008.
FEAR, G.; KOMARNYTSKY, S.; RASKIN, I. Proteases inhibitors and their peptidomimetic derivates as potential drugs. Pharmacology and Therapeutics, v.113, p.354-368, 2007.
GÖRANSSON, U.; SVANGAND, E.; CLAESON, P.; BOHLIN, L. Novel strategies for isolation and characterization of cyclotides: the discovery of bioactive macrocyclic plant polypeptides in the Violaceae. Current Protein Peptide Scence, v.5, p.317-29, 2004.
HEAL, W.P.; WICKRAMASINGHE, S.R.; TATE, E.W. Activity based chemical proteomics: profiling proteases as drug targets. Current Drug Discovery Technology, v.5, p.200-212, 2008.
KENNEDY, A.R. Chemopreventive agents: Protease inhibitors. Pharmacology and Therapeutics, v.78, p.167-209, 1998.
KROWARSCH, D.; CIERPICKI, T.; JELEN, F.; OTLEWSKI, J. Canonical protein inhibitors of serine proteases. Cellular Molecular Life Science, v.60, p.2427-2444, 2003.
KUESTER, D.; LIPPERT, H.; ROESSNER, A.; KRUEGER, S. The cathepsin family and their role in colorectal cancer. Pathology Research Practise, v.204, p.491-500, 2008.
LASKOWSKI, M.J.; KATO, I. Protein inhibitors of proteinases. Annual Review of Biochemistry, v.49, p.593-626, 1980.
LASKOWSKI, M.J.; QASIM, M.A. What can the structures of enzyme-inhibitor complexes tell us about the structures of enzyme substrate complexes? Biochimica et Biophysics Acta, v.1477, p.324-337, 2000.
LOSSO, J.N. The biochemical and functional food properties of the bowman-birk inhibitor. Critical Revision of Food Science Nutritional. v.48, n.1, p.94-118, 2008.
NEURATH, H. Proteolytic processing and physiological regulation. Trends Biochemistry Science, v.14, p.268-271, 1989.
OTLEWSKI J.; JELEN, F.; ZAKRZEWSKA, M.; OLESY, A. The many faces of protease-protein inhibitor Interaction. The EMBO Journal, v.24, p.1303-1310, 2005.
OTLEWSKI, J.; KROWARSCH, D. Squash inhibitor of serine proteinases. Acta Biochimica Polonica, v.43, p.431-444, 1996.
POWERS, J.C.; ASGIAN, J.L.; EKICI, O.; JAMES, K.E. Irreversible inhibitors of serine, cysteine and threonine proteases. Chemical Reviews, v.102, p. 4639-4750, 2002.
QI, R.F; SONG, Z.W.; CHI, C.W. Structural Features and Molecular Evolution of Bowman-Birk Protease Inhibitors and Their Potential Application. Acta Biochimica and Biophysics Sinica, v.37, p.283-292, 2005.
RAWLIINGS, N.D.; BARRET, A.J. Evolutionary families of peptidases. Biochememistry Journal, v.290, p.205-218, 1993.
RAWLINGS, N.D.; TOLLE, D.P.; BARRETT, A.J. Evolutionary families of peptidase inhibitors. Biochemistry Journal, v.378, p.705-716, 2004.
RAWLINGS, N.D.; MORTON, F.R. The MEROPS batch BLAST: A tool to detect peptidases and their non-peptidase homologues in a genome. Biochimie, v.90, p.243-259, 2008.
ROBERTS, T.H.; HEJGAARD, J. Serpins in plants and green algae. Function and Integrative Genomics, v.8, p.1-27, 2008.
SAMPATH, A., PADMANABHAN, R. Molecular targets for flavivirus drug discovery. Antiviral Research, v.81, p.124-132, 2008.
SCHECHTER I.; BERGER, A. Biochemistry and Biophysics Research Communications, v. 27, p.157-160, 1967.
SILVA-LOPEZ, R.E.; MORGADO-DÍAZ, J.A.; CHÁVEZ, M.A.; GIOVANNI-DE-SIMONE, S. (2007) Effects of serine protease inhibitors on viability and morphology of Leishmania (Leishmania) amazonensis promastigotes. Parasitology Research, v.101, p.1627-1635, 2007.
WILLIAMS, B.; DICKMAN, M. Plant programmed cell death: can’t live with it; can’t live without it. Molecular Plant Pathology, v.9, p.531-44, 2008.
WOODS, S.; FARRALL, A.; PROCKO, C.; WHITELAW, M.L. The bHLH/Per-Arnt-Sim transcription factor SIM2 regulates muscle transcript myomesin2 via a novel, non-canonical E-box sequence. Nucleic Acids Research, v.36, p.3716-27, 2008.
YANG, L.; TADA, Y.; YAMAMOTO, M.P.; ZHAO, H.; YOSHIKAWA, M.; TAKAIWA, F.A Transgenic rice seed accumulating an anti-hypertensive peptide reduces the blood pressure of spontaneously hypertensive rats. FEBS Letters, v.580, p.3315-3320, 2006.
YOSHIZAKI, L.; TRONCOSO, M.F.; LOPES, J.L.S.; HELLMAN, U.; BELTRAMINI, L.M.; WOLFENSTEIN-TODEL, C. Calliandra selloi Macbride trypsin inhibitor: Isolation, characterization, stability, spectroscopic analyses. Phytochemistry, v.68, p.2625-2634, 2007.
ZHANG, Y.; KOUZUMA, Y.; MIYAJI, T.; YONEKURA, M. Purification, characterization, and cDNA cloning of a Bowman-Birk type trypsin inhibitor from Apios americana Medikus tubers. Bioscience Biotechnology and Biochemistry, v.72, p.171-178, 2008.